Mechanizmy degradacji RNA w mitochondriach
Agnieszka.Rajda
pon., 14/02/2022 – 16:17
Mitochondria to struktury w żywej komórce, które są odpowiedzialne za wiele kluczowych procesów, w tym za dostarczanie niezbędnej do życia komórki energii. U ludzi defekty mitochondriów prowadzą do bardzo poważnych chorób, często o podłożu neurologicznym. Unikalną cechą mitochondriów jest to, że posiadają swój własny materiał genetyczny (kwas deoksyrybonukleinowy – DNA) oraz maszynerię do jego odczytywania. Pierwszym etapem tego odczytywania jest przepisywanie DNA do kwasu rybonukleinowego – RNA. RNA jest następnie odczytywane w procesie produkcji białek. Zapewnienie odpowiedniej ilości białek jest niezbędne do prawidłowego funkcjonowania mitochondriów. Głównym sposobem ustalania tej ilości jest regulowanie poziomu RNA, które koduje białka. W mitochondriach dzieje się to przede wszystkim przez proces rozkładania (degradacji) RNA. Dlatego degradacja ta odgrywa tak ważną rolę w prawidłowym funkcjonowaniu mitochondriów.
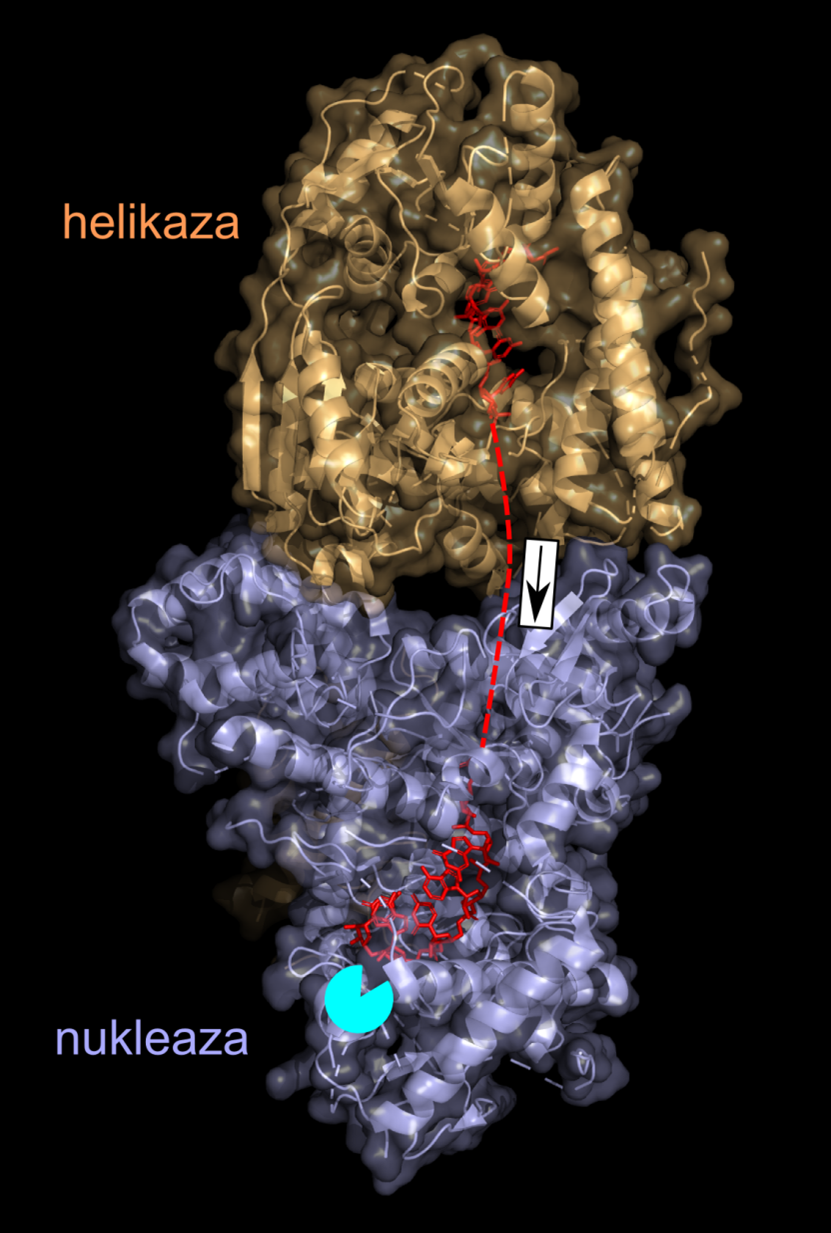 Komputerowa wizualizacja atomowej struktury przestrzennej degradosomu z mitochondriów drożdży. Helikaza jest pokazana na pomarańczowo, nukleaza – na niebiesko, a nić RNA – na czerwono. Fragment nie zaobserwowany bezpośrednio w strukturach oznaczono linią przerywaną. Kierunek przesuwania RNA pokazuje strzałka, a miejsce, gdzie odcinane są nukleotydy z końca RNA – turkusowe koło. Autor: Marcin Nowotny.
Komputerowa wizualizacja atomowej struktury przestrzennej degradosomu z mitochondriów drożdży. Helikaza jest pokazana na pomarańczowo, nukleaza – na niebiesko, a nić RNA – na czerwono. Fragment nie zaobserwowany bezpośrednio w strukturach oznaczono linią przerywaną. Kierunek przesuwania RNA pokazuje strzałka, a miejsce, gdzie odcinane są nukleotydy z końca RNA – turkusowe koło. Autor: Marcin Nowotny.
Celem naszego projektu było uzyskanie jak najbardziej kompletnego obrazu mechanizmów degradacji RNA w mitochondriach. Chcieliśmy je poznać zarówno na poziomie bardziej ogólnym, w kontekście całych mitochondriów, jak i bardzo szczegółowym – na poziomie poszczególnych atomów, które wchodzą w skład zaangażowanych w ten proces białek.
W ramach projektu zidentyfikowaliśmy nowe białka, które biorą udział w regulacji poziomu RNA. Wśród nich jest ludzkie białko MTRES1. Pełni ono funkcję ochronną, przeciwdziała obniżeniu ilości funkcjonalnych RNA, gdy mitochondria znajdują się w trudnych warunkach. Badaliśmy również inne białko z mitochondriów o nazwie GRSF1. Pokazaliśmy, że współdziała ono z degradosomem, główną maszynerią mitochondrialną odpowiedzialną za rozkład RNA. Odkryliśmy, że współdziałanie to dotyczy RNA, które zawiera wiele guanozyn (liter G informacji genetycznej). Cząsteczki takiego RNA przyjmują specjalną, bardzo stabilną strukturę przestrzenną. Pokazaliśmy, że rolą GRSF1 jest destabilizowanie tych specjalnych struktur, aby ułatwić ich rozkładanie (praca opublikowana w „Nature Communications” w 2018 r.).
Maszyneria degradosomu składa się z dwóch białek. Jedno z nich (zwane helikazą) przesuwa nić RNA. Drugie, zwane nukleazą, odcina pojedyncze nukleotydy (litery informacji genetycznej) z jednego końca nici RNA. Aby dogłębnie zrozumieć ten proces, użyliśmy metody krystalografii białek, w celu określenia atomowej struktury degradosomu z drożdży. Struktura pokazała, że helikaza i nukleaza wiążą się ze sobą tworząc kompaktową maszynę, w której helikaza wpycha RNA do kanału w nukleazie. Na końcu kanału znajduje się miejsce, w którym zachodzi reakcja chemiczna odcinania nukleotydów. Oba białka tworzą więc ściśle skoordynowaną efektywną maszynę do rozkładania RNA (praca opublikowana w „Nature Communications” w 2018 r.). Badaliśmy również bardziej ogólnie funkcjonowanie degradosomu drożdży. Pokazaliśmy, że jego rolą jest zapewnienie, aby w mitochondriach obecne były funkcjonalne RNA (w tym kodujące białka), a niefunkcjonalne RNA były usuwane.
Istotną częścią projektu było również opracowanie metod komputerowych, które pozwalają na analizę danych dotyczącej sekwencji liter informacji genetycznej w RNA z mitochondriów, a także analizę tych regionów RNA, które oddziałują z białkami. Zaletą nowo opracowanych metod jest to, że mogą być one użyte do trudnych zestawów danych, również takich, w których znajduje się duża różnorodność sekwencji. Realizacja tego projektu dostarczyła ważnych informacji o przetwarzaniu RNA w mitochondriach człowieka oraz organizmów prostszych (drożdży). Pozwala to lepiej zrozumieć funkcjonowanie tych kluczowych dla komórki struktur. Dane dla komórek ludzkich są również podstawą do zrozumienia poważnych chorób wynikających z defektów ludzkich mitochondriów i tworzenia strategii ich leczenia.
Laboratorium Struktury Białka w MiBMiK. Fot. Marcin Nowotny
Od 2008 r. kierownik laboratorium w Międzynarodowym Instytucie Biologii Molekularnej i Komórkowej. Laureat ERC Starting Grant (2011). W latach 2003-2008 odbył staż podoktorski w National Institutes of Health w USA. W 2002 r. uzyskał stopień doktora w Instytucie Biologii Doświadczalnej PAN.
Członkowie zespołu:
Dr hab. Roman Szczęsny
Od 2017 r. kierownik grupy w Instytucie Biochemii i Biofizyki PAN. W latach 2010-2018 odbył staż podoktorski na Uniwersytecie Warszawskim oraz w Instytucie Biochemii i Biofizyki PAN. W 2009 r. uzyskał stopień doktora w Instytucie Biochemii i Biofizyki PAN.
Prof. dr hab. Paweł Golik
Dyrektor Instytutu Genetyki i Biotechnologii na Wydziale Biologii Uniwersytetu Warszawskiego. W latach 2000-2002 odbył staż podoktorski na Emory University w Atlancie, USA. W 1999 r. uzyskał stopień doktora na Wydziale Biologii Uniwersytetu Warszawskiego we współpracy z Centre de Génétique Moléculaire, CNRS w Gif-sur-Yvette we Francji.
Dr hab. Bartosz Wilczyński, prof. UW
Od 2021 r. profesor uczelni na Uniwersytecie Warszawskim, na Wydziale Matematyki, Informatyki i Mechaniki. Od 2011 r. – adiunkt. W latach 2008-2011 odbył staż podoktorski w European Molecular Biology Laboratory w Heidelbergu w Niemczech. W 2008 r. uzyskał doktorat w Instytucie Matematycznym PAN.
Na zdjęciu od lewej: Marcin Nowotny, Bartosz Wilczyński, Roman Szczęsny, Paweł Golik. Fot. Daria Goś
Source: NCN
